> Методы > Разное > Измерение концентрации на спектрофотометре.
Спектрофотометрическое измерение концентрации DNA( RNA)(для DU7500).
- предел измерения прибора ~0.1µg/ml, т.е. на анализ требуется > 0.01µg. Удобнее пользоваться "маленькой" (100µl) ячейкой. Чтобы кривизна поверхности жидкости не влияла на измерения лучше брать объем образца равным 130µl.
- С[µg/ml] = А260 x K
- На отношение A260/A280(1.8-1.9 - весьма чистая DNA, 1.9-2.0 – RNA) имеет смысл обращать внимание только если измерение проводится в буферном растворе (например, ТЕ) при нейтральном pH.
- При работе с pDNA добавление к чистой плазмидной DNA до 2х кратного избытка RNA приводит к практически линейному сдвигу A260/A280 от 1.9 до 2.1.
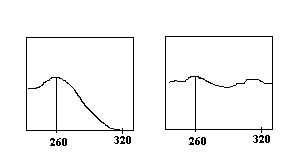
- Точность измерения падает при слишком больших и при слишком маленьких поглощениях:
Измерение концентрации DNA/RNA.
Если для анализа 1µl образца разбавляется в 130µl буфера, то коэффициент пересчета K1:130OD260 в µg:
|
* - для олигонуклеотидов коэффициент сильно зависит от состава.
Если совсем строго: {20mM Na фосфат, 0.1M NaCl}. Такие низкосолевые буферы как Tris 10-100mM, pH7.5-9.0; KHPO4100mM, pH8.2, дают очень сходные результаты.
Измерения в воде приводят к отклонениям до 14% и заниженному A260/A280.